Bild: Deutsches Krebsforschungszentrum, Heidelberg
Biophysiker, Biologen und Mediziner suchen seit längerem nach Methoden der individualisierten Tumor-Therapie mit weniger unerwünschten Nebenwirkungen im Vergleich zur heutigen Radio- und Chemotherapie. Ein Team unter Leitung von Dr. Christopher J. Tape am weltweit renommierten University College London (UCL) scheint nun diesem Traum einen bedeutenden Schritt näher gekommen zu sein. In einem am 17. Februar 2020 im Fachmagazin „nature methods“ online veröffentlichten Beitrag beschreiben sie eine Methode der Analyse des Verhaltens von Millionen unterschiedlicher Zellen im Innern von im Labor gezüchteten Mini-Tumoren.
Man wusste schon, dass Tumorzellen die Wachstumssignale gesunder Zellen imitieren, damit sie unkontrolliert wuchern können. Die von Tape und Mitarbeitern entwickelte Methode erlaubt es nun, das Verhalten von Krebszellen, Immunzellen und normalen Zellen simultan und bis aufs kleinste Detail zu analysieren. Die Forscher benutzten dafür künstlich geschaffene dreidimensionale Mini-Tumore (Organoide). Diese werden aus Darmtumor-Stammzellen in Kollagen gezüchtet. Gegenüber zweidimensionalen Zellkulturen aus identischen Zellen haben diese den Vorteil, alle relevanten Zelltypen zu enthalten. Um das Verhalten der verschiedenen Zelltypen individuell beobachten zu können, entwickelten die Londoner Forscher eine neue Massenspektroskopie-Plattform. Die verschiedenen Zelltypen zeichnen sich durch charakteristische Post-Translational Modifications (PTM) aus. Diese Proteine werden mit Antikörpern, die mit Schwermetallen kenntlich gemacht wurden, markiert. Auf diese Weise wurden über 40 PTM-Typen charakterisiert. Das erlaubt es den Forschern, mit High-Throughput-Techniken verschiedene Regelkreise aufzudecken und die Wirkung verschiedener Medikamente oder Immuntherapien zu verfolgen. So können die Therapien auf die individuellen Eigenheiten der Patienten zugeschnitten werden.
Qin, X., Sufi, J., Vlckova, P. et al. Cell-type-specific signaling networks in heterocellular organoids. Nat Methods (2020). https://doi.org/10.1038/s41592-020-0737-8
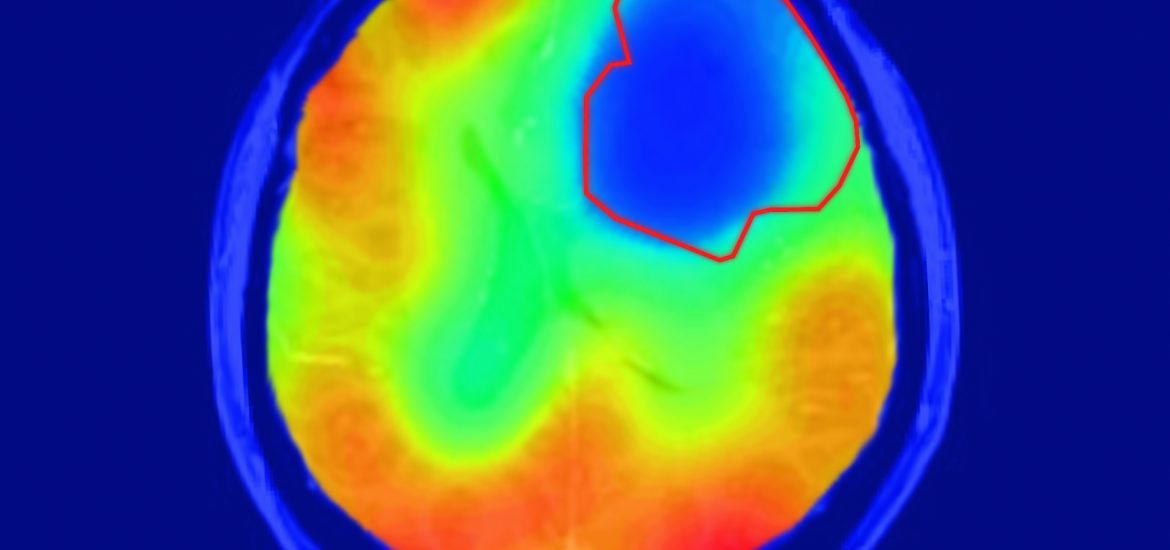
Einen anderen Weg, Krebs-Therapien zielgerichteter und damit effizienter zu machen, haben Forscher des Deutschen Krebsforschungszentrums DKFZ in Heidelberg eingeschlagen. Die von ihnen am 18. Februar 2020 im Fachjournal „Radiology“ veröffentlichte Methode soll vor allem der Diagnostik und Charakterisierung schwer zugänglicher Hirntumore dienen. Sie entsannen sich der schon in den 1920er Jahren vom deutschen Arzt und Biochemiker Otto Heinrich Warburg aufgestellten Krebs-Theorie. Warburg hatte beobachtet, dass sich in den Tumorzellen viel Milchsäure (Laktat) ansammelt. Laktat ist das Endprodukt des wenig effektiven anaeroben Stoffwechsels, bei dem Kohlenhydrate und andere energiereiche Nahrungsbestandteile nur vergärt, aber nicht anschließend über den Citratzyklus und die Atmungskette der Mitochondrien mit Sauerstoff verbunden werden. Krebszellen sind dadurch charakterisiert, dass sie die Gärung auch dann bevorzugen, wenn ihnen genügend Sauerstoff zur Verfügung steht. Es galt also eine Methode zu finden, mit der Zellen, die nur vergären, von atmenden Zellen sicher unterschieden werden können.
Der Arzt und Physiker Daniel Paech und seine Mitarbeiter fanden die Lösung in einer Verbindung der Magnetresonanztomografie (MRT) mit der Einatmung des stabilen Sauerstoff-Isotops 17-O2. Dieses findet sich in Form geringer Spuren auch in der normalen Atemluft. Für die Diagnostik muss das Spurengas künstlich angereichert werden. Haben die Probanden die mit 17-O2 angereicherte Luft eingeatmet, dann erscheinen alle Gewebe mit einem hohen Sauerstoff-Umsatz auf dem entsprechend programmierten Computerbildschirm hell, während Tumore als dunkle Flecken erscheinen.
Daniel Paech, Armin M. Nagel, Miriam N. Schultheiss, Reiner Umathum, Sebastian Regnery, Moritz Scherer, Antje Wick, MD Tanja Platt, Wolfgang Wick, Martin Bendszus, Andreas Unterberg, Heinz-Peter Schlemmer, Mark E. Ladd, Sebastian C. Niesporek: Quantitative dynamic oxygen-17 MRI at 7-T for cerebral oxygen metabolism in glioma
Radiology 2020, DOI: https://doi.org/10.1148/radiol.2020191711